
資訊中心
作者:香雪生命科學(xué)--盧華
發(fā)布時(shí)間:2021-04-29
閱讀:12134
大家伙可能都聽(tīng)說(shuō)過(guò),在2003年經(jīng)過(guò)多個(gè)國家的多年努力,完成了宏大的人類(lèi)基因組計劃,該工程通過(guò)測序確定了人類(lèi)染色體中30億個(gè)堿基對組成的DNA序列,被譽(yù)為生命科學(xué)研究的“登月計劃”。
對整個(gè)基因組(生物體中所有的DNA)進(jìn)行測序是一項復雜的任務(wù)。它需要將基因組DNA先分解成許多較小的片段,然后對這些小片段進(jìn)行測序,接著(zhù)將這些序列組裝成完整的DNA序列。 隨著(zhù)技術(shù)不斷發(fā)展和改進(jìn),目前的全基因組測序速度更快,成本更低。本文給大家介紹經(jīng)典的測序方法——Sanger測序,然后簡(jiǎn)要介紹新一代測序技術(shù)
Sanger測序:鏈終止法
Sanger測序技術(shù)是由英國生物化學(xué)專(zhuān)家Sanger教授和其同事于1977年開(kāi)發(fā),該方法測定的DNA片段一般不超過(guò)900個(gè)堿基。在人類(lèi)基因組計劃中,Sanger測序用于確定人類(lèi)DNA中許多相對較小片段的序列,但這些片段不一定小于900 個(gè)堿基,研究人員可以利用多輪Sanger測序沿著(zhù)每個(gè)片段進(jìn)行測序,再根據重疊部分對片段進(jìn)行比對,組裝較大DNA區域的序列,最終組裝整個(gè)染色體序列。
Sanger測序原理
Sanger測序涉及制備靶DNA區域的多個(gè)拷貝,它的組成類(lèi)似于生物體中復制DNA或體外復制DNA的聚合酶鏈反應(PCR)所需的成分:DNA聚合酶,引物,四種脫氧核苷酸(dNTP)以及待測序的DNA模板。
當然,Sanger測序反應也有其獨特的成分,這也是其核心原理:四種不同染料標記的雙脫氧核苷酸(ddNTP)。相比常規的脫氧核苷酸,雙脫氧核苷酸糖環(huán)的3’碳原子上缺少羥基。因此,在DNA合成過(guò)程中一旦雙脫氧核苷酸添加到鏈中,將無(wú)法正常形成磷酸二酯鍵,進(jìn)而使DNA合成反應終止。該DNA鏈也就以染料標記的雙脫氧核苷酸結尾,可以利用凝膠電泳和顯影技術(shù)根據條帶位置確定待測DNA片段的序列。

圖一 Sanger測序
Sanger測序基本過(guò)程
在DNA合成反應體系中加入一定比例帶有染料標記的ddNTP,其數量要比普通核苷酸少得多。反應完成后,體系中包含了不同長(cháng)度的DNA片段,終止于待測DNA序列的每個(gè)核苷酸位置,并且片段的末端被帶有特定染料的ddNTP標記。
然后,將片段通過(guò)一根含有凝膠基質(zhì)的毛細管,該過(guò)程也稱(chēng)為凝膠電泳。短片段能夠快速地穿過(guò)凝膠,而長(cháng)片段相對移動(dòng)較慢。當每個(gè)片段越過(guò)管子末端時(shí),都會(huì )被激發(fā)光照射,從而檢測出ddNTP上附著(zhù)的染料。
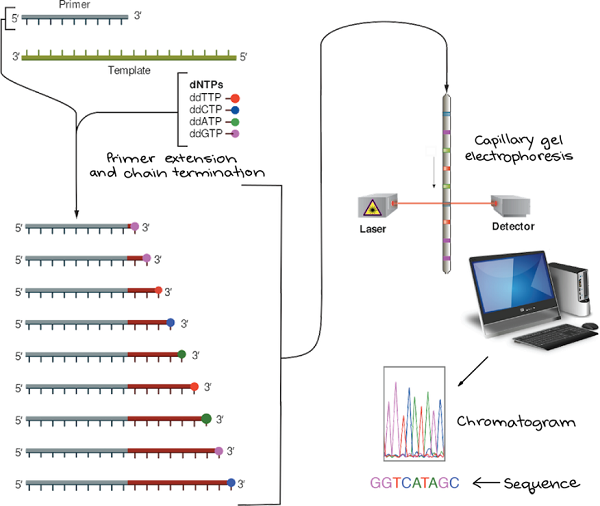
圖二 測序流程
最小的片段(在引物后僅插入了一個(gè)核苷酸)首先穿過(guò)終點(diǎn)線(xiàn),然后是第二個(gè)最小的片段(在引物后插入了兩個(gè)核苷酸),依此類(lèi)推。因此,檢測器記錄的數據由一系列熒光強度峰組成,可以根據檢測器上一個(gè)接一個(gè)記錄的染料顏色,從色譜圖中讀取待測DNA的序列。
用途和局限性
Sanger測序可為較長(cháng)的DNA片段(最多約900個(gè)堿基對)提供高質(zhì)量的測定,主要用于對單個(gè)DNA片段進(jìn)行測序,如細菌生產(chǎn)的質(zhì)粒或者是PCR克隆的DNA。
但是,對于大型項目,如整個(gè)基因組或宏基因組(微生物菌群的“集體基因組”)測序,Sanger測序昂貴且效率低下。對于這類(lèi)任務(wù),新的高通量測序技術(shù)更快,更便宜。
Next-generation sequencing
最新的DNA測序技術(shù)統稱(chēng)為下一代測序(NGS),也稱(chēng)高通量測序。有許多使用不同技術(shù)的新一代測序方法,但大多數具有相同的特點(diǎn)。相比傳統的Sanger測序,NGS支持多個(gè)測序反應同時(shí)進(jìn)行,測序反應體系小,可以在芯片上一次完成多組反應等優(yōu)點(diǎn)。但是,其測序長(cháng)度相對要短一些,通常為50-700個(gè)堿基。
從概念上講,下一代測序有點(diǎn)像同時(shí)運行大量微小的Sanger測序反應。由于這種并行化和小規模,與Sanger測序相比,新一代測序技術(shù)可以更快,更便宜地對大量DNA進(jìn)行測序。例如,在2001年,對人類(lèi)基因組進(jìn)行測序的成本接近1億美元。但到了2015年只需要大約1000美元!
為什么快速廉價(jià)的測序很重要?對DNA序列進(jìn)行常規測序的能力為生物學(xué)研究和生物醫學(xué)應用開(kāi)辟了新的可能性。例如,低成本的測序是朝著(zhù)精準醫學(xué)邁出的一步,也就是說(shuō),根據他或她基因組中的變異基因,可以針對個(gè)體差異量身定制個(gè)性化醫療方案。
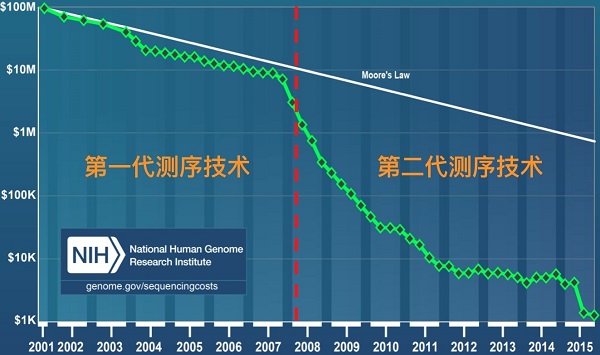
圖三 測序成本比較
目前,第二代高通量測序技術(shù)以Roche公司的454平臺、ABI公司的SOLiD平臺和illumina公司的Solexa平臺為代表。454平臺采用了焦磷酸測序法,是首個(gè)被發(fā)明的商用二代測序儀。SOLiD平臺是由獨特的連接酶測序法發(fā)展而來(lái),檢測過(guò)程中兩個(gè)堿基確定一個(gè)熒光信號,相當于一次能檢測兩個(gè)堿基,因此該方法也被稱(chēng)為兩堿基測序法。Solexa平臺是現在應用最廣泛的二代測序平臺,作為測序界的巨鱷,占據了全球70%以上的測序儀器市場(chǎng)份額。其測序原理是基于可逆性鏈終止物和橋式PCR等專(zhuān)有技術(shù)的邊合成邊測序法(SBS)。
第二代測序技術(shù)常用的應用包括小型全基因組測序(微生物和病毒)、靶向基因測序(基于擴增子、基因panel)、轉錄組測序(總RNA-Seq、mRNA-Seq和基因表達圖譜分析)、甲基化測序以及16S宏基因組測序等。
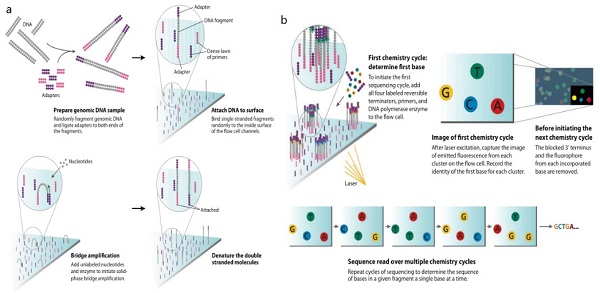
圖四 SBS流程
近幾年,隨著(zhù)關(guān)鍵性技術(shù)的突破和革命性的發(fā)明,誕生了第三代高通量測序技術(shù)。第三代測序技術(shù)也稱(chēng)為單分子測序技術(shù),根據測序技術(shù)原理主要分為兩大陣營(yíng):第一陣營(yíng)是單分子熒光測序技術(shù),代表性技術(shù)為Pac Bio公司的SMRT技術(shù)。該技術(shù)為了實(shí)現實(shí)時(shí)測序,利用零模波導孔技術(shù)避免了反應體系中游離的熒光標記核苷酸產(chǎn)生的強大熒光背景,同時(shí)開(kāi)發(fā)出新的核苷酸熒光標記方法,將傳統的堿基熒光標記改為標記磷酸基團,使得標記物會(huì )在DNA合成過(guò)程中自動(dòng)脫落,大大提高了DNA聚合酶的活性和測序讀長(cháng)。第二陣營(yíng)為Oxford Nanopore Technologies公司的單分子納米孔測序技術(shù)。與以往的光信號檢測手段不同,該方法采用電信號檢測,不依賴(lài)DNA聚合酶,因此理論上能夠做到超長(cháng)的測序讀長(cháng)。
相比第二代測序,第三代測序最大特點(diǎn)是單分子測序,過(guò)程中不涉及PCR過(guò)程,因此能夠有效避免PCR擴增的偏向性和引入突變,缺失等系統性錯誤。另外,第三代測序還能直接對DNA進(jìn)行甲基化測序分析,不需要事先對基因組進(jìn)行Bisulfite處理。但是,目前第三代測序平臺仍存在一些不足,需進(jìn)一步改進(jìn)和完善,如測序成本和錯誤率較高等。

圖五 MinlON測序儀
單細胞測序
自2013年,《Nature Methods》將單細胞測序評為年度技術(shù),單細胞測序逐漸成為熱點(diǎn)技術(shù)。為什么呢?一方面是因為測序成本大大降低,使得NGS已經(jīng)成為實(shí)驗室里的家常便飯。另一方面,細胞作為生命結構和功能的基本單位,以往的測序樣品往往是組織樣本或細胞群,檢測結果是多個(gè)細胞的平均值,很容易掩蓋了異質(zhì)性細胞的信息。單細胞水平測序能夠使測序結果更加全面,多層次,復雜程度更高。
簡(jiǎn)單來(lái)說(shuō),單細胞測序就是在測序前增加一步單細胞分離和標記的過(guò)程,常用的分離技術(shù)包括激光捕獲顯微切割、流式細胞分選、微孔捕獲技術(shù)和微流控技術(shù)等。目前,單細胞測序主要應用于空間基因表達、單細胞染色質(zhì)轉座酶可接近性分析(ATAC)、單細胞免疫分析和單細胞基因表達等。通過(guò)以上應用我們可以探索細胞間異質(zhì)性,鑒定新的靶點(diǎn)和生物標志物,分析配對的全長(cháng)B細胞或T細胞受體、抗原特異性、細胞表面蛋白表達和染色質(zhì)開(kāi)放性等。
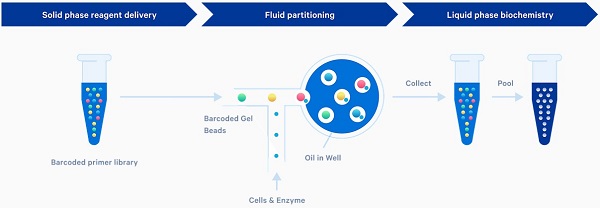
圖六 10× Genomics的Chromium技術(shù)
小結
目前,NGS已經(jīng)被廣泛的應用于臨床醫學(xué)和基礎研究中,作為破解生命遺傳信息的重要手段,測序技術(shù)還在飛快的發(fā)展進(jìn)化。相信在未來(lái),測序技術(shù)能夠幫助我們更全面的,更深層次的理解生命中的各種奧秘和疾病,甚至幫助我們攻克癌癥。
參考文獻:
1. Lewis, T. (2013, April 14) Human genome project marks 10th anniversary. In LiveScience.
2. National Human Genome Research Institute. (2016) DNA sequencing costs. In Large-scale genome sequencing and analysis centers (LSAC).
3. Obenrader, S. (2003) The Sanger method. In Sarah Obenrader: Molecular biology.
4. Reece, J.B., Urry, L.A., and Cain, M.L. (2011) Dideoxy chain termination method for sequencing DNA. In Campbell biology (10th ed., p. 410) San Francisco, CA: Pearson.
5. Thermo Fisher Scientific. (2016) Sanger sequencing method. In Sanger sequencing.
6. Shendure J, Ji H. Next-generation DNA sequencing. Nat Biotechnol. 2008;26(10):1135-1145. doi:10.1038/nbt1486
7. Zhang Z, Shen J, Wang H, et al. Effects of Graphene Nanopore Geometry on DNA Sequencing. J Phys Chem Lett. 2014;5(9):1602-1607. doi:10.1021/jz500498c
8. Mardis ER. Next-generation DNA sequencing methods. Annu Rev Genomics Hum Genet. 2008;9:387-402. doi:10.1146/annurev.genom.9.081307.164359
9. Metzker ML. Sequencing technologies - the next generation. Nat Rev Genet. 2010; 11(1):31-46. doi:10.1038/nrg2626
10. Niedringhaus TP, Milanova D, Kerby MB, Snyder MP, Barron AE. Landscape of next-generation sequencing technologies. Anal Chem. 2011;83(12):4327-4341. doi:10.1021/ ac2010857
11. Method of the year 2013. Nat Methods. 2014;11(1):1. doi:10.1038/nmeth.2801